Тег SNP - Википедия - Tag SNP
А тег SNP является представителем однонуклеотидный полиморфизм (SNP) в области генома с высоким нарушение равновесия по сцеплению который представляет собой группу SNP, называемую гаплотип. Можно идентифицировать генетические вариации и ассоциации с фенотипами без генотипирования каждого SNP в хромосомной области. Это сокращает затраты и время на картирование областей генома, связанных с заболеванием, поскольку устраняет необходимость изучения каждого отдельного SNP. Тег SNP полезен в полногеномные ассоциации SNP в котором генотипированы сотни тысяч SNP по всему геному.
Вступление
Нарушение равновесия сцепления

Два места говорят, что находятся в равновесие сцепления (LE) если их наследование - самостоятельное событие. Если аллели в этих локусах наследуются неслучайно, то мы говорим, что они находятся в нарушение равновесия по сцеплению (LD). LD чаще всего вызывается физическим сцеплением генов. Когда два гена наследуются на одной и той же хромосоме, в зависимости от их расстояния и вероятности рекомбинации между локусами они могут иметь высокую LD. Однако LD также может наблюдаться из-за функциональных взаимодействий, когда даже гены из разных хромосом могут совместно давать эволюционно выбранный фенотип или могут влиять на жизнеспособность потенциального потомства.
В семьях LD является самым высоким из-за наименьшего количества событий рекомбинации (наименьшее количество событий мейоза). Это особенно верно между инбредными линиями. В популяциях LD существует из-за отбора, физической близости генов, которая вызывает низкие скорости рекомбинации, или из-за недавнего скрещивания или миграции. На уровне популяции процессы, влияющие на неравновесие по сцеплению, включают: генетическая связь, эпистатический естественный отбор, скорость рекомбинации, мутация, генетический дрейф, случайная вязка, генетический автостоп и поток генов.[2]
Когда группа SNP наследуется вместе из-за высокого LD, имеется тенденция к избыточной информации. Выбор тега SNP в качестве представителя этих групп снижает количество избыточности при анализе частей генома, связанных с признаками / заболеваниями.[3] Области генома с высоким LD, содержащие определенный набор SNP, которые наследуются вместе, также известны как гаплотипы. Следовательно, теги SNP являются репрезентативными для всех SNP внутри гаплотипа.
Гаплотипы
Выбор SNP меток зависит от гаплотипов, присутствующих в геноме. Большинство технологий секвенирования предоставляют генотипическую информацию, а не гаплотипы, то есть они предоставляют информацию о конкретных присутствующих основаниях, но не предоставляют фазовую информацию (на какой конкретной хромосоме появляется каждое из оснований).[4] Определение гаплотипов можно проводить молекулярными методами (Аллель-специфическая ПЦР, Гибриды соматических клеток ). Эти методы определяют, какой аллель присутствует на какой хромосоме, путем разделения хромосом перед генотипированием. Они могут быть очень трудоемкими и дорогими, поэтому методы статистического вывода были разработаны как менее затратный и автоматизированный вариант. Эти программные пакеты статистического вывода используют алгоритмы экономии, максимального правдоподобия и байесовские алгоритмы для определения гаплотипов. Недостатком статистического вывода является то, что часть предполагаемых гаплотипов может быть неверной.[5]
Популяционные различия
Когда гаплотипы используются для полногеномных ассоциативных исследований, важно отметить изучаемую популяцию. Часто разные популяции имеют разные модели LD. Одним из примеров дифференциации моделей является население африканского происхождения по сравнению с населением европейского и азиатского происхождения. Поскольку люди произошли в Африке и распространились в Европе, а затем на азиатском и американском континентах, африканские популяции являются наиболее генетически разнообразными и имеют меньшие области LD, в то время как европейские и азиатские популяции имеют более крупные области LD из-за эффект основателя. Когда паттерны LD различаются в популяциях, SNP могут быть диссоциированы друг с другом из-за изменений в блоки гаплотипов. Это означает, что теговые SNP, как представители блоков гаплотипов, уникальны в популяциях, и при проведении ассоциативных исследований следует учитывать популяционные различия.[6]
Заявление
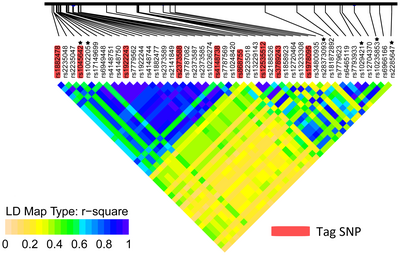
GWAS
Почти каждая черта имеет как генетическое влияние, так и влияние окружающей среды. Наследственность - это доля фенотипической изменчивости, унаследованная от наших предков. Ассоциативные исследования используются для определения генетическое влияние на фенотипическое представление. Хотя в основном они используются для сопоставления болезней с областями генома, их можно использовать для сопоставления наследственности любого фенотипа, такого как рост, цвет глаз и т. Д.
Полногеномные исследования ассоциации (GWAS) использовать однонуклеотидные полиморфизмы (SNP) определить генетические ассоциации с клиническими состояниями и фенотипическими признаками.[8] Они свободны от гипотез и используют полногеномный подход для исследования признаков путем сравнения большой группы людей, которые выражают фенотип, с большой группой людей, которые этого не делают. Конечная цель GWAS - определить генетические факторы риска, которые можно использовать для прогнозирования того, кто подвергается риску заболевания, каковы биологические основы восприимчивости к заболеванию, и создать новые стратегии профилактики и лечения.[1] В Национальный институт исследования генома человека и Европейский институт биоинформатики издает Каталог GWAS, каталог опубликованных полногеномных исследований ассоциаций, в которых подчеркиваются статистически значимые ассоциации между сотнями SNP с широким диапазоном фенотипов.[9]

Из-за большого количества возможных вариантов SNP (более 149 млн на июнь 2015 г. [10][11]) по-прежнему очень дорого секвенировать все SNP. Вот почему GWAS использует настраиваемые массивы (Чипы SNP) для генотипирования только подмножества вариантов, идентифицированных как tag snps. Большинство GWAS используют продукты двух основных платформ генотипирования. В Affymetrix Платформа печатает ДНК-зонды на стеклянном или силиконовом чипе, которые гибридизуются с конкретными аллелями в образце ДНК. В Иллюмина платформа использует технологию на основе шариков, с более длинными последовательностями ДНК и обеспечивает лучшую специфичность.[1] Обе платформы способны генотипировать более миллиона тегов SNP с использованием готовых или пользовательских ДНК олигонуклеотидов.
Полногеномные исследования основаны на гипотеза об общем заболевании-распространенном варианте (CD / CV) в котором говорится, что на общие расстройства влияют общие генетические вариации. Размер эффекта (пенетрантность ) общих вариантов должно быть меньше по сравнению с вариантами, обнаруженными при редких заболеваниях. Это означает, что общий SNP может объяснить только небольшую часть дисперсии из-за генетических факторов и что на общие заболевания влияют несколько общих аллелей с небольшим размером эффекта. Другая гипотеза заключается в том, что общие заболевания вызываются редкими вариантами, которые синтетически связанный к распространенным вариантам. В этом случае сигнал, производимый GWAS, является косвенной (синтетической) ассоциацией между одним или несколькими редкими причинными вариантами при неравновесном сцеплении. Важно понимать, что это явление возможно при выборе группы для тегов SNP. Когда обнаруживается, что заболевание связано с гаплотипом, некоторые SNP в этом гаплотипе будут иметь синтетическую связь с заболеванием. Чтобы точно определить причинные SNP, нам нужно большее разрешение при выборе блоков гаплотипов. Поскольку технологии полногеномного секвенирования быстро меняются и становятся все менее дорогими, вполне вероятно, что они заменят современные технологии генотипирования, обеспечивая разрешение, необходимое для точного определения причинных вариантов.
HapMap
Поскольку секвенирование всего генома людей по-прежнему является непомерно дорогостоящим, международный проект HapMap был создан с целью сопоставить геном человека с группами гаплотипов (блоками гаплотипов), которые могут описывать общие образцы генетической изменчивости человека. Путем сопоставления всего генома с гаплотипами можно идентифицировать SNP-метки, представляющие блоки гаплотипов, проверенные генетическими исследованиями. Важным фактором, который следует учитывать при планировании генетического исследования, является частота и риск определенных аллелей. Эти факторы могут различаться в разных популяциях, поэтому в проекте HapMap использовались различные методы секвенирования для обнаружения и каталогизации SNP из разных наборов популяций. Первоначально в рамках проекта были секвенированы индивиды из популяции йоруба африканского происхождения (YRI), жители штата Юта западноевропейского происхождения (CEU), неродственные индивиды из Токио, Япония (JPT) и неродственные ханьцы из Пекина, Китай (CHB). Недавно их наборы данных были расширены, чтобы включить другие группы населения (11 групп). [1]
Выбор и оценка
Шаги по выбору тега SNP
Выбор максимально информативных SNP тегов является NP полная проблема. Однако могут быть разработаны алгоритмы, обеспечивающие приближенное решение в пределах погрешности.[12] Критерии, необходимые для определения каждого алгоритма выбора SNP тегов, следующие:
- Определите область для поиска - алгоритм попытается найти SNP тегов в окрестности N (t) целевого SNP t
- Определите метрику для оценки качества тегов - метрика должна измерять, насколько хорошо целевой SNP t может быть предсказан с использованием набора его соседей N (t), т.е. насколько хорошо теговый SNP как представитель SNP в окрестности N (t) может предсказать целевой SNP t . Его можно определить как вероятность того, что целевой SNP t имеет разные значения для любой пары гаплотипов i и j, где значение SNP s также отличается для одних и тех же гаплотипов. Информативность метрики можно представить в терминах теории графов, где каждый SNP s представлен в виде графа Gs, узлы которого являются гаплотипами. Gs имеет ребро между узлами (i, j) тогда и только тогда, когда значения s различны для гаплотипов Hi, Hj.[12]
- Вывести алгоритм поиска репрезентативных SNP - цель алгоритма - найти минимальное подмножество тегов SNP, выбранных с максимальной информативностью между каждым тегом SNP с каждым другим целевым SNP
- Проверить алгоритм
Выбор функции
Методы выбора функций делятся на две категории: методы фильтрации и методы-оболочки. Алгоритмы фильтрации - это общие алгоритмы предварительной обработки, которые не предполагают использование определенного метода классификации. Алгоритмы оболочки, напротив, «оборачивают» выбор функций вокруг определенного классификатора и выбирают подмножество функций на основе точности классификатора с использованием перекрестной проверки.[13]
Метод выбора признаков, подходящий для выбора тегов SNP, должен иметь следующие характеристики:
- хорошо масштабируется для большого количества SNP;
- не требуют явной маркировки классов и не должны предполагать использование определенного классификатора, потому что классификация не является целью маркировки выбора SNP;
- позволяют пользователю выбирать разное количество тегов SNP для разного количества допустимой потери информации;
- имеют сопоставимые характеристики с другими методами, удовлетворяющими трем первым условиям.
Алгоритмы выбора
Было предложено несколько алгоритмов выбора тегов SNP. Первый подход был основан на измерении качества наборов SNP и поиске подмножеств SNP, которые являются небольшими, но достигают высокого значения определенной меры. Изучение каждого подмножества SNP для поиска подходящих с вычислительной точки зрения выполнимо только для небольших наборов данных.
Другой подход использует анализ главных компонент (PCA) чтобы найти подмножества SNP, улавливающих большую часть дисперсии данных. Метод скользящих окон используется для многократного применения PCA к коротким хромосомным областям. Это уменьшает количество производимых данных, а также не требует экспоненциального времени поиска. Тем не менее, невозможно применить метод PCA к большим наборам хромосомных данных, поскольку он сложен в вычислительном отношении.[13]
Наиболее часто используемый подход, блочный метод, использует принцип неравновесия по сцеплению, наблюдаемый внутри блоков гаплотипов.[12] Было разработано несколько алгоритмов для разделения хромосомных областей на блоки гаплотипов, которые основаны на разнообразие гаплотипов, LD, тест с четырьмя гаметами и информационная сложность и теги SNP выбираются из всех SNP, которые принадлежат этому блоку. Основное предположение в этом алгоритме состоит в том, что SNP двуаллельный.[14] Главный недостаток заключается в том, что определение блоков не всегда однозначно. Несмотря на то, что существует список критериев для формирования блоков гаплотипов, единого мнения по нему нет. Кроме того, выбор SNP тегов на основе локальной корреляции игнорирует межблочные корреляции.[12]
В отличие от блочного подхода, безблочный подход не зависит от блочной структуры. Частота SNP и скорости рекомбинации, как известно, варьируются в зависимости от генома, и в некоторых исследованиях сообщалось, что расстояния LD намного превышают указанные максимальные размеры блоков. Установка строгой границы для соседства нежелательна, и безблочный подход ищет теговые SNP глобально. Для этого есть несколько алгоритмов. В одном алгоритме SNP без тегов представлены как логические функции тегов SNP и теория множеств методы используются для уменьшения пространства поиска. Другой алгоритм ищет подмножества маркеров, которые могут поступать из непоследовательных блоков. Из-за близости маркера пространство поиска сокращается.[13]
Оптимизация
Поскольку число генотипированных людей и количество SNP в базах данных растет, выбор тегов SNP занимает слишком много времени для вычисления. Чтобы повысить эффективность метода выбора тега SNP, алгоритм сначала игнорирует двуаллельные SNP, а затем сжимает длину (номер SNP) матрицы гаплотипов, группируя сайты SNP с одинаковой информацией. Сайты SNP, которые разделяют гаплотипы на одну группу, называются избыточными сайтами. Сайты SNP, которые содержат различную информацию в пределах блока, называются неизбыточными сайтами (NRS). Для дальнейшего сжатия матрицы гаплотипов алгоритм должен найти SNP тегов, чтобы можно было различить все гаплотипы матрицы. Используя идею совместного разбиения, предоставляется эффективный алгоритм выбора SNP тегов.[14]
Проверка точности алгоритма
В зависимости от того, как выбираются SNP тегов, в процессе перекрестной проверки использовались разные методы прогнозирования. Метод машинного обучения был использован для прогнозирования оставленного гаплотипа. Другой подход предсказал аллели нетегирующего SNP n из теговых SNP, которые имели самый высокий коэффициент корреляции с n. Если обнаруживается один высококоррелированный тег SNP t, аллели присваиваются таким образом, чтобы их частоты совпадали с частотами аллелей t. Когда несколько SNP с тегами имеют одинаковый (высокий) коэффициент корреляции с n, общий аллель n имеет преимущество. Легко видеть, что в этом случае метод прогнозирования хорошо согласуется с методом выбора, который использует PCA на матрице коэффициентов корреляции между SNP.[13]
Есть и другие способы оценки точности метода выбора SNP тега. Точность можно оценить с помощью показателя качества R2, который представляет собой меру связи между истинным количеством копий гаплотипов, определенных для полного набора SNP, и прогнозируемым количеством копий гаплотипов, где прогноз основан на подмножестве помеченных SNP. Эта мера предполагает диплоидные данные и явный вывод гаплотипов из генотипов.[13]
Другой метод оценки, предложенный Клейтоном, основан на измерении разнообразия гаплотипов. Разнообразие определяется как общее количество различий во всех парных сравнениях между гаплотипами. Разница между парой гаплотипов - это сумма различий по всем SNP. Мера разнообразия Клейтона может использоваться для определения того, насколько хорошо набор тегов SNP дифференцирует различные гаплотипы. Этот показатель подходит только для блоков гаплотипов с ограниченным разнообразием гаплотипов, и неясно, как его использовать для больших наборов данных, состоящих из нескольких блоков гаплотипов.[13]
В некоторых недавних работах оцениваются алгоритмы выбора SNP с тегами на основе того, насколько хорошо SNP с тегами можно использовать для прогнозирования SNP без тегов. Точность прогнозирования определяется с помощью перекрестной проверки, такой как исключение одного или удержание. При перекрестной проверке с исключением по одному для каждой последовательности в наборе данных алгоритм запускается на остальной части набора данных, чтобы выбрать минимальный набор тегирующих SNP.[13]
Инструменты
Tagger
Tagger - это веб-инструмент, доступный для оценки и выбора SNP тегов из генотипических данных, таких как Международный проект HapMap. В нем используются парные методы и подходы с использованием мультимаркерных гаплотипов. Пользователи могут загрузить данные генотипа HapMap или формат родословной, и будут рассчитаны паттерны неравновесия сцепления. Параметры Tagger позволяют пользователю указать хромосомные ориентиры, которые указывают интересующие области в геноме для выбора SNP тегов. Затем программа создает список SNP тегов и их статистические тестовые значения, а также отчет о покрытии. Он разработан Полом де Баккером в лабораториях Дэвида Альтшулера и Марка Дейли в Центре генетических исследований человека. Массачусетская больница общего профиля и Гарвардская медицинская школа, на Broad Institute.[15]
CLUSTAG и WCLUSTAG
В бесплатном программном обеспечении CLUSTAG и WCLUSTAG содержатся алгоритмы кластера и покрытия набора для получения набора тегов SNP, которые могут представлять все известные SNP в хромосомной области. Программы реализованы на Java, и они могут работать как на платформе Windows, так и в среде Unix. Они разработаны СИО-ИОНГ АО и другие. в Университете Гонконга.[16][17]
Смотрите также
- Международный проект HapMap
- Полногеномное исследование ассоциации
- Однонуклеотидный полиморфизм
- Нарушение равновесия сцепления
Рекомендации
- ^ а б c d Буш, Уильям С .; Мур, Джейсон Х .; Льюиттер, Фрэн; Канн, Марисель (27 декабря 2012 г.). «Глава 11: Общегеномные исследования ассоциации». PLOS вычислительная биология. 8 (12): e1002822. Дои:10.1371 / journal.pcbi.1002822. ЧВК 3531285. PMID 23300413.
- ^ ван дер Верф, Юлиус. «Основы сцепления и картирования генов» (PDF). Получено 30 апреля 2014.
- ^ Левонтин, Р. (1988). «О показателях гаметического неравновесия». Генетика. 120 (3): 849–852. ЧВК 1203562. PMID 3224810.
- ^ Гальперин, Е .; Kimmel, G .; Шамир, Р. (16 июня 2005 г.). «Выбор метки SNP в данных генотипа для максимальной точности предсказания SNP». Биоинформатика. 21 (Приложение 1): i195 – i203. Дои:10.1093 / биоинформатика / bti1021. PMID 15961458.
- ^ Кроуфорд, Дана К .; Никерсон, Дебора А. (2005). «Определение и клиническое значение гаплотипов». Ежегодный обзор медицины. 56 (1): 303–320. Дои:10.1146 / annurev.med.56.082103.104540. PMID 15660514.
- ^ Тео, ГГ; Sim, X (апрель 2010 г.). «Паттерны неравновесия сцепления в разных популяциях: последствия и возможности для липид-ассоциированных локусов, выявленных в результате полногеномных исследований ассоциации». Текущее мнение в липидологии. 21 (2): 104–15. Дои:10.1097 / MOL.0b013e3283369e5b. PMID 20125009.
- ^ Шоу, Вэйхуа; Ванга, Дажи; Чжан, Кайюэ; Ван, Бейлан; Ван, Чжимин; Ши, Цзиньсю; Хуанг, Вэй; Хуан, Цинъян (26 сентября 2012 г.). «Общегеновая характеристика локусов общих количественных признаков для экспрессии мРНК ABCB1 в нормальных тканях печени в китайской популяции». PLOS ONE. 7 (9): e46295. Дои:10.1371 / journal.pone.0046295. ЧВК 3458811. PMID 23050008.
- ^ Welter, D .; MacArthur, J .; Morales, J .; Burdett, T .; Холл, П .; Junkins, H .; Klemm, A .; Flicek, P .; Manolio, T .; Hindorff, L .; Паркинсон, Х. (6 декабря 2013 г.). "Каталог NHGRI GWAS, кураторский ресурс ассоциаций SNP-признаков". Исследования нуклеиновых кислот. 42 (D1): D1001 – D1006. Дои:10.1093 / nar / gkt1229. ЧВК 3965119. PMID 24316577.
- ^ Витте, Джон С .; Хоффманн, Томас Дж. (2011). «Полигенное моделирование общегеномных ассоциативных исследований: приложение к раку простаты и груди». OMICS: журнал интегративной биологии. 15 (6): 393–398. Дои:10.1089 / omi.2010.0090. ЧВК 3125548. PMID 21348634.
- ^ Статистика данных dbSNP. Национальный центр биотехнологической информации (США). 2005 г.
- ^ "Резюме dbSNP".
- ^ а б c d Тарво, Алекс. «Учебное пособие по тегированию гаплотипов» (PDF). Получено 1 мая 2014.
- ^ а б c d е ж грамм Phuong, TM; Lin, Z; Альтман, РБ (апрель 2006 г.). «Выбор SNP с помощью выбора характеристик». Журнал биоинформатики и вычислительной биологии. 4 (2): 241–57. CiteSeerX 10.1.1.128.1909. Дои:10.1109 / csb.2005.22. PMID 16819782.
- ^ а б Чен, WP; Hung, CL; Tsai, SJ; Лин, Ю.Л. (2014). «Новые и эффективные алгоритмы выбора тегов SNP». Биомедицинские материалы и инженерия. 24 (1): 1383–9. Дои:10.3233 / BME-130942. PMID 24212035.
- ^ "Tagger". Получено 1 мая 2014.
- ^ "КЛУСТАГ". Получено 16 мая 2014.
- ^ "WCLUSTAG". Получено 16 мая 2014.
