Phi X 174 - Phi X 174
| Вирус эшерихии ΦX174 | |
|---|---|
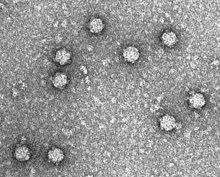 | |
| Электронная микрофотография фага ΦX174 | |
| Классификация вирусов | |
| (без рейтинга): | Вирус |
| Царство: | Моноднавирия |
| Королевство: | Sangervirae |
| Тип: | Phixviricota |
| Класс: | Malgrandaviricetes |
| Порядок: | Petitvirales |
| Семья: | Микровирусы |
| Род: | Синшеймервирус |
| Виды: | Вирус эшерихии ΦX174 |
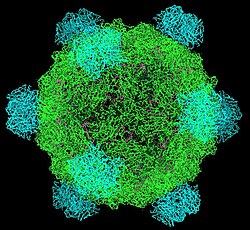

В фи X 174 (или ΦX174) бактериофаг представляет собой одноцепочечную ДНК (оцДНК ) вирус, который поражает кишечная палочка, и первая основанная на ДНК геном быть упорядоченным. Эта работа была завершена Фред Сэнгер и его команда в 1977 году.[2] В 1962 г. Уолтер Фирс и Роберт Зиншеймер уже продемонстрировали физическую ковалентно замкнутую кольцевость ДНК ΦX174.[3] Лауреат Нобелевской премии Артур Корнберг использовали ΦX174 в качестве модели, чтобы сначала доказать, что ДНК, синтезированная в пробирке с помощью очищенных ферментов, может производить все свойства природного вируса, открывая эру синтетическая биология.[4][5] В 1972-1974 гг. Джерард Гурвиц, Сью Викнер, и Рид Уикнер с сотрудниками идентифицировали гены, необходимые для производства ферментов, катализирующих превращение одноцепочечной формы вируса в двухцепочечную репликативную форму.[6] В 2003 г. Крейга Вентера группа, что геном ΦX174 был первым, который был полностью собран in vitro из синтезированных олигонуклеотидов.[7] Частица вируса ΦX174 также была успешно собрана. in vitro.[8] В 2012 году было показано, насколько высоко перекрывающийся геном может быть полностью распакован и по-прежнему работать.[9]
Геном
Эта бактериофаг имеет [+] смысл круговой одноцепочечный ДНК геном 5,386 нуклеотиды.[1] Геном GC-контент составляет 44% и 95% нуклеотидов принадлежат кодирующим генам. Из-за сбалансированной базовой структуры генома она используется в качестве контрольной ДНК для секвенсоров Illumina.
Гены
ΦX174 кодирует 11 генов, названных последовательными буквами алфавита в том порядке, в котором они были обнаружены, за исключением A *, который является альтернативным стартовым кодоном в больших генах A. Считается, что только гены A * и K несущественны, хотя есть некоторые сомнения относительно A *, потому что его стартовый кодон может быть изменен на ATT, но не на любую другую последовательность.[10] Теперь известно, что АТТ все еще способен продуцировать белок.[11] в пределах Кишечная палочка и поэтому этот ген не может быть несущественным.
Фаг ΦX174 использовался, чтобы попытаться установить отсутствие неоткрытой генетической информации с помощью подхода «доказательства путем синтеза».[12]
Транскриптом
В 2020 году транскриптом из ΦX174.[13] Примечательными особенностями транскриптома ΦX174 являются ряд до четырех относительно слабых промоторов, последовательно включенных до четырех Rho-независимых (внутренних) терминаторов и одного Rho-зависимого терминатора.
Белки
ΦX174 кодирует 11 белки.
| Протеин | Копии | Функция[14] |
|---|---|---|
| А | -- | Ники РФ ДНК для инициации репликация катящегося круга; лигирует концы линейной ДНК фага с образованием одноцепочечной кольцевой ДНК |
| А * | -- | Подавляет репликацию ДНК клетки-хозяина; блокирует суперинфицирующий фаг; не важно |
| B | 60 дюйм прокапсид | Белок внутреннего каркаса, участвующий в сборке прокапсида |
| C | -- | Упаковка ДНК |
| D | 240 в прокапсиде | Белок внешнего каркаса, участвующий в сборке прокапсида |
| E | -- | Клетка-хозяин лизис |
| F | 60 в вирионе | Основной белок капсида |
| г | 60 в вирионе | Основной протеин шипа |
| ЧАС | 12 в вирионе | Пилотный белок ДНК (или второстепенный спайковый белок) |
| J | 60 в вирионе | Связывается с новой одноцепочечной ДНК фага; сопровождает ДНК фага в прокапсид |
| K | -- | Оптимизирует размер пакета; не важно |
Протеом
Недавно сообщалось об идентификации всех белков ΦX174 с помощью масс-спектрометрии.[15]
Цикл заражения
Заражение начинается, когда G-белок связывается с липополисахариды на поверхности бактериальной клетки-хозяина. H-белок (или ДНК-пилотный белок) направляет вирусный геном через бактериальную мембрану Кишечная палочка бактерии[16] скорее всего через предсказанный N-терминал трансмембранный домен спираль.[17] Однако стало очевидно, что белок H является многофункциональным белком.[18] Это единственный вирусный капсид белок ΦX174 не имеет кристаллической структуры по нескольким причинам. Имеет низкое содержание ароматических веществ и высокое глицин содержание, что делает структуру белка очень гибкой и, кроме того, отдельные атомы водорода (группа R для глицинов) трудно обнаружить в кристаллографии белка. Кроме того, белок H индуцирует лизис бактериального хозяина в высоких концентрациях, поскольку предсказанная N-концевая трансмембранная спираль легко протыкает бактериальную стенку. От биоинформатика, этот белок содержит четыре предсказанных спиральная катушка домены, которые имеют значительную гомологию с известными факторами транскрипции. Кроме того, было установлено, что de novo Белок H был необходим для оптимального синтеза других вирусных белков.[19] Мутации в белке H, которые предотвращают включение вируса, можно преодолеть, если доставить избыточное количество белка B, внутреннего каркасного белка.
ДНК выбрасывается через гидрофильный канал в 5-кратной вершине.[20] Понятно, что белок H находится в этой области, но экспериментальные данные не подтвердили его точное местоположение. Попав внутрь бактерии-хозяина, репликация генома [+] оцДНК происходит через отрицательный смысл ДНК промежуточный. Это происходит по мере того, как суперспирали фагового генома и вторичная структура, образованная такой суперспирализацией, привлекают примосома белковый комплекс. Он один раз перемещается по геному и синтезирует [-] оцДНК из положительного исходного генома. [+] Геномы оцДНК для упаковки в вирусы создаются по механизму катящегося круга. Это механизм, с помощью которого двухцепочечный суперспиральный геном разрывается на положительной цепи кодируемым вирусом A-белком, что также привлекает бактериальный ДНК-полимераза (DNAP) к сайту расщепления. DNAP использует отрицательную цепь в качестве матрицы для создания положительной смысловой ДНК. По мере того как он перемещается по геному, он вытесняет внешнюю цепь уже синтезированной ДНК, которая немедленно покрывается SSBP белки. Белок A расщепляет полный геном каждый раз, когда он распознает исходную последовательность.
Поскольку белок D является наиболее распространенным транскриптом гена, он является наиболее распространенным белком в вирусном прокапсиде. Точно так же транскрипты генов для F, J и G более многочисленны, чем для H, поскольку стехиометрия для этих структурных белков 5: 5: 5: 1. Примосомы представляют собой белковые комплексы, которые прикрепляют / связывают фермент. геликаза по шаблону. Примосомы дают праймеры РНК для синтеза ДНК цепям.
Использует
Эволюция
Он использовался как модельный организм во многих эволюционных экспериментах.[21]
Биотехнологии
ΦX174 регулярно используется как положительный контроль в Секвенирование ДНК из-за относительно небольшого размера генома по сравнению с другими организмами, его относительно сбалансированное содержание нуклеотидов - около 23% G, 22% C, 24% A и 31% T, то есть 45% G + C и 55% A + T , см. присоединение NC_001422.1[1] за его последовательность длиной 5386 нуклеотидов. Иллюмина в приборах секвенирования используется ΦX174 в качестве положительного контроля,[22] а один цикл секвенирования Illumina может охватить геном ΦX174 несколько миллионов раз, что делает этот геном, вероятно, наиболее сильно секвенированным геномом в истории.[нужна цитата ]
ΦX174 также используется для проверки сопротивления средства индивидуальной защиты к вирусам, передающимся с кровью.[23]
ΦX174 также был модифицирован для включения пептидного дисплея (фагового дисплея) из G-белка вирусного капсида.[24]
Синтетическая биология
Геном ΦX174 был первым фагом, клонированным в дрожжах,[25] что обеспечивает удобный сухой док для модификации генома.[26] ΦX174 был также первым геномом, который был полностью декомпрессирован с удалением всех перекрытий генов. Эффект этих изменений привел к значительному снижению привязанности к хозяину, нарушению регуляции экспрессии белков и чувствительности к теплу.[27]
Смотрите также
использованная литература
- ^ а б c Энтеробактерии фаг phiX174 Sensu lato, полный геном. «Полный геном: образец NC_001422», Национальный центр биотехнологической информации. Проверено 30 января, 2016.
- ^ Сэнгер Ф., Air GM, Баррелл Б.Г., Браун Н.Л., Колсон А.Р., Фиддес, Калифорния, Хатчисон, Калифорния, Слокомб П.М., Смит М. (февраль 1977 г.). «Нуклеотидная последовательность ДНК бактериофага phi X174». Природа. 265 (5596): 687–95. Bibcode:1977Натура.265..687С. Дои:10.1038 / 265687a0. PMID 870828. S2CID 4206886.
- ^ Fiers W, Sinsheimer RL (октябрь 1962 г.). «Структура ДНК бактериофага ΦX174. III. Ультрацентробежные доказательства кольцевой структуры». Журнал молекулярной биологии. 5 (4): 424–34. Дои:10.1016 / S0022-2836 (62) 80031-X. PMID 13945085.
- ^ Национальная библиотека медицинских профилей в науке. Документы Артура Корнберга. «Создание жизни в пробирке», 1959-1970 гг. ссылка на сайт[неосновной источник необходим ]
- ^ Гулиан М., Корнберг А., Зиншеймер Р.Л. (декабрь 1967 г.). «Ферментативный синтез ДНК, XXIV. Синтез ДНК инфекционного фага phi-X174». Труды Национальной академии наук Соединенных Штатов Америки. 58 (6): 2321–8. Bibcode:1967PNAS ... 58.2321G. Дои:10.1073 / pnas.58.6.2321. JSTOR 58720. ЧВК 223838. PMID 4873588.
- ^ Сью Викнер и Джерард Гурвиц (1974) "Преобразование вирусной ДНК Phi X174 в двухцепочечную форму очищенной кишечная палочка Белки ". Proc Natl Acad Sci USA 71(10):4122-4124.
- ^ Smith HO, Hutchison CA, Pfannkoch C, Venter JC (декабрь 2003 г.). «Создание синтетического генома путем сборки всего генома: бактериофаг phiX174 из синтетических олигонуклеотидов». Труды Национальной академии наук Соединенных Штатов Америки. 100 (26): 15440–5. Bibcode:2003ПНАС..10015440С. Дои:10.1073 / pnas.2237126100. JSTOR 3149024. ЧВК 307586. PMID 14657399.
- ^ Cherwa JE, Organtini LJ, Ashley RE, Hafenstein SL, Fane BA (сентябрь 2011 г.). «В VITRO ASSEMBLY прокапсида ΦX174 из олигомеров внешних каркасных белков и промежуточных продуктов ранней пентамерной сборки». Журнал молекулярной биологии. 412 (3): 387–96. Дои:10.1016 / j.jmb.2011.07.070. PMID 21840317.
- ^ Яшке П.Р., Либерман Е.К., Родригес Дж., Сьерра А., Энди Д. (декабрь 2012 г.). «Полностью декомпрессированный геном синтетического бактериофага ΦX174, собранный и заархивированный в дрожжах». Вирусология. 434 (2): 278–84. Дои:10.1016 / j.virol.2012.09.020. PMID 23079106.
- ^ Baas, P.D .; Liewerink, H .; van Teeffelen, H.A .; van Mansfeld, A.D .; van Boom, J. H .; Янс, Х. С. (22 июня 1987 г.). «Изменение стартового кодона ATG белка A бактериофага phi X174 на кодон ATT дает жизнеспособный фаг, указывающий на то, что белок A не является существенным для воспроизводства phi X174». Письма FEBS. 218 (1): 119–125. Дои:10.1016 / 0014-5793 (87) 81030-х. ISSN 0014-5793. PMID 2954853. S2CID 24174007.
- ^ Хехт, Ариэль; Глазго, Джефф; Jaschke, Paul R .; Bawazer, Lukmaan A .; Мансон, Мэтью С .; Кокран, Дженнифер Р .; Энди, Дрю; Салит, Марк (20 апреля 2017 г.). «Измерения инициации трансляции со всех 64 кодонов в E. coli». Исследования нуклеиновых кислот. 45 (7): 3615–3626. Дои:10.1093 / нар / gkx070. ЧВК 5397182. PMID 28334756.
- ^ Jaschke, Paul R .; Dotson, Gabrielle A .; Hung, Kay S .; Лю, Дайан; Энди, Дрю (26 ноября 2019). «Окончательная демонстрация полноты аннотации генома путем синтеза». Труды Национальной академии наук. 116 (48): 24206–24213. Дои:10.1073 / pnas.1905990116. ЧВК 6883844. PMID 31719208.
- ^ Логель, Доминик Й .; Яшке, Пол Р. (август 2020 г.). «Карта транскрипции бактериофага ϕX174 с высоким разрешением». Вирусология. 547: 47–56. Дои:10.1016 / j.virol.2020.05.008. PMID 32560904.
- ^ Фейн Б.А., Брентлингер К.Л., Берч А.Д., Чен М., Хафенштейн С., Мур Э., Новак С.Р., Учияма А. (2006). "ɸX174 et al., Микровирусы". В календаре R (ред.). Бактериофаги (2-е изд.). Нью-Йорк: Oxford Univ. Нажмите. п. 130. ISBN 978-0195148503.
- ^ Райт, Брэдли У .; Руан, Цзюаньфанг; Molloy, Mark P .; Яшке, Пол Р. (12 октября 2020 г.). «Модуляризация генома показывает, что топология перекрывающихся генов необходима для эффективного размножения вирусов». Синтетическая биология ACS. Дои:10.1021 / acssynbio.0c00323. ISSN 2161-5063.
- ^ Язвински С.М., Линдберг А.А., Корнберг А. (1975). «Липополисахаридный рецептор бактериофагов ΦX174 и S13». Вирусология. 66 (1): 268–282. Дои:10.1016 / 0042-6822 (75) 90197-х. PMID 1094681.
- ^ Тушнади Г.Е., Симон I (сентябрь 2001 г.). «Сервер прогнозирования трансмембранной топологии HMMTOP». Биоинформатика. 17 (9): 849–50. Дои:10.1093 / биоинформатика / 17.9.849. PMID 11590105.
- ^ Черва Дж. Э., Янг Л. Н., Фейн Б. А. (март 2011 г.). «Разделение функций многофункционального белка: выделение мутанта экспериментального белка ДНК, который влияет на морфогенез частиц». Вирусология. 411 (1): 9–14. Дои:10.1016 / j.virol.2010.12.026. PMID 21227478.
- ^ Рубойанес М.В., Чен М., Дубрава М.С., Черва Д.Е., Фане Б.А. (октябрь 2009 г.). «Экспрессия пилотных белков ДНК с N-концевой делецией ингибирует ранние стадии репликации phiX174». Журнал вирусологии. 83 (19): 9952–6. Дои:10.1128 / JVI.01077-09. ЧВК 2748053. PMID 19640994.
- ^ Маккенна Р., Ся Д., Виллингманн П., Илаг Л.Л., Кришнасвами С., Россманн М.Г., Олсон Н.Х., Бейкер Т.С., Инкардона Н.Л. (январь 1992 г.). «Атомная структура одноцепочечной ДНК бактериофага phi X174 и ее функциональные последствия». Природа. 355 (6356): 137–43. Bibcode:1992Натура.355..137М. Дои:10.1038 / 355137a0. ЧВК 4167681. PMID 1370343.
- ^ Вичман HA, Браун CJ (август 2010 г.). «Экспериментальная эволюция вирусов: Microviridae как модельная система». Философские труды Лондонского королевского общества. Серия B, Биологические науки. 365 (1552): 2495–501. Дои:10.1098 / rstb.2010.0053. ЧВК 2935103. PMID 20643739.
- ^ «Использование PhiX Control для циклов секвенирования HiSeq®». Illumina. Архивировано из оригинал 9 января 2019 г.. Получено 8 января 2019.
- ^ «СИЗ-Инфо - Стандартные детали». wwwn.cdc.gov. Получено 8 февраля 2019.
- ^ Christakos, Kristofer J .; Чепмен, Дженис А .; Fane, Bentley A .; Кампос, Самуэль К. (январь 2016 г.). «PhiXing-it, отображение чужеродных пептидов на бактериофаге ΦX174». Вирусология. 488: 242–248. Дои:10.1016 / j.virol.2015.11.021. ЧВК 6191337. PMID 26655242.
- ^ Jaschke, Paul R .; Либерман, Эрика К .; Родригес, Джон; Сьерра, Адриан; Энди, Дрю (декабрь 2012 г.). «Полностью декомпрессированный геном синтетического бактериофага øX174, собранный и заархивированный в дрожжах». Вирусология. 434 (2): 278–284. Дои:10.1016 / j.virol.2012.09.020. ISSN 0042-6822. PMID 23079106.
- ^ Андо, Хироки; Лемир, Себастьян; Пирес, Диана П .; Лу, Тимоти К. (сентябрь 2015 г.). «Разработка модульных вирусных матриксов для целевого редактирования популяции бактерий». Сотовые системы. 1 (3): 187–196. Дои:10.1016 / j.cels.2015.08.013. ЧВК 4785837. PMID 26973885.
- ^ Райт, Брэдли У .; Руан, Цзюаньфанг; Molloy, Mark P .; Яшке, Пол Р. (12 октября 2020 г.). «Модуляризация генома показывает, что топология перекрывающихся генов необходима для эффективного размножения вирусов». Синтетическая биология ACS. Дои:10.1021 / acssynbio.0c00323. ISSN 2161-5063.
внешние ссылки
- Гудселл, Дэвид (февраль 2000 г.). «Бактериофаг phiX174». Молекула месяца. RCSB-PDB.
