PA клан протеаз - PA clan of proteases
| PA клан протеазы | |
|---|---|
 | |
| Идентификаторы | |
| Символ | Нет данных |
| Pfam клан | CL0124 |
| ИнтерПро | IPR009003 |
| SCOP2 | 50494 / Объем / СУПФАМ |
| Мембранома | 319 |
В Клан ПА (пролевые игры смешанного нуклеофила, надсемейство А) - самая большая группа протеазы с общим происхождением, как определено структурная гомология. Члены имеют химотрипсин -подобная складка и подобные протеолиз механизмы, но могут иметь идентичность <10%. В клан входят оба цистеин и сериновые протеазы (разные нуклеофилы ).[1][2] Протеазы клана PA можно найти в растения,[3] животные,[3] грибы,[3] эубактерии,[4] археи[5][6] и вирусы.[2]
Обычное использование каталитическая триада для гидролиза несколькими кланами протеаз, включая клан PA, представляет собой пример конвергентная эволюция.[7] Различия в каталитической триаде внутри клана PA также являются примером дивергентная эволюция из активные сайты в ферментах.[2]
История
В 1960-е гг. сходство последовательностей нескольких протеаз указывает на их эволюционное родство.[8] Они были сгруппированы в химотрипсиноподобные сериновые протеазы[9] (теперь называется Семья S1 ). Поскольку структуры этих и других протеаз были решены с помощью Рентгеновская кристаллография в 1970-х и 80-х годах было замечено, что несколько вирусных протеаз, таких как Протеаза вируса травления табака показал структурная гомология несмотря на отсутствие заметного сходства последовательностей и даже наличия другого нуклеофила.[2][10][11] Основываясь на структурной гомологии, a надсемейство был определен и позже назван кланом ПА ( МЕРОПЫ система классификации). По мере того как решается больше структур, к суперсемейству клана PA добавляется больше семейств протеаз.[12][13]
Этимология
В п относится к протеазы смешанного нуклеофила. В А указывает, что это был первый такой клан, который был идентифицирован (существуют также кланы PB, PC, PD и PE).[1]
Структура
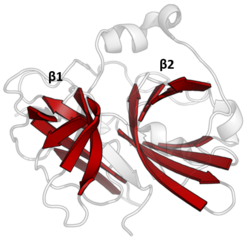
Несмотря на сохранение всего лишь 10% идентичности последовательностей, члены клана PA, изолированные от вирусов, прокариот и эукариот, демонстрируют структурная гомология и может быть выровнен по структурному сходству (например, с ДАЛИ ).[3]
Двойной β-ствол
Все протеазы клана PA имеют два основных мотива β-стволы с ковалентным катализом, выполняемым кислотно-гистидин-нуклеофилом каталитическая триада мотив. Цилиндры расположены перпендикулярно друг другу, а гидрофобные остатки удерживают их вместе в качестве каркаса ядра для фермента. Остатки триады разделяются между двумя барабанами так, чтобы катализ происходит в их интерфейсе.[14]
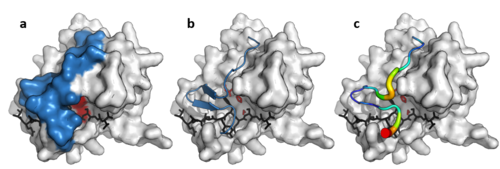
Петля вирусной протеазы
В дополнение к двойному β-цилиндрическому ядру некоторые вирусные протеазы (такие как Протеаза TEV ) долго, гибкий С-концевая петля, которая образует крышку, которая полностью покрывает субстрат и создает туннель связывания. Этот туннель содержит набор плотно связывающих карманов, так что каждая боковая цепь пептида-субстрата (от P6 до P1 ’) связана в комплементарном сайте (от S6 до S1’), а специфичность обеспечивается большой площадью контакта между ферментом и субстратом.[11] И наоборот, клеточные протеазы, в которых отсутствует эта петля, такие как трипсин иметь более широкий специфичность.
Эволюция и функции
Каталитическая активность
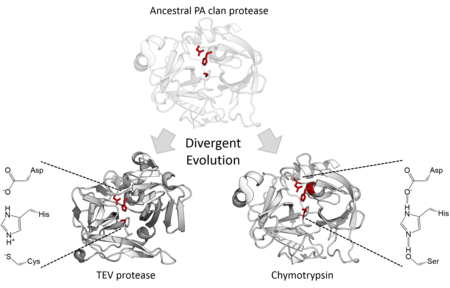
Структурная гомология указывает на то, что члены клана PA произошли от общего предка одной и той же группы. Хотя протеазы клана PA используют каталитическую триаду, выполняющую двухступенчатую нуклеофильный катализ,[7] некоторые семьи используют серин как нуклеофил тогда как другие используют цистеин.[2] Таким образом, суперсемейство является крайним примером дивергентная эволюция ферментов поскольку в ходе эволюционной истории основной каталитический остаток фермента менялся в разных семьях.[15] Помимо структурного сходства, направленная эволюция было показано, что он способен превращать цистеиновую протеазу в активную сериновую протеазу.[16] Все протеазы клеточного клана PA являются сериновые протеазы, однако есть как серин, так и цистеиновая протеаза семейства вирусных протеаз.[7] Большинство из них эндопептидазы, за исключением семейства S46. экзопептидазы.[17][18]
Биологическая роль и субстратная специфичность
Помимо дивергенции в их основном каталитическом аппарате, протеазы клана PA также обнаруживают широкую дивергентную эволюцию функций. Члены клана PA можно найти в эукариоты, прокариоты и вирусы и включают в себя широкий спектр функций. У млекопитающих некоторые участвуют в свертывание крови (например. тромбин ) и поэтому обладают высокой субстратной специфичностью, а также пищеварение (например. трипсин ) с широкой субстратной специфичностью. Несколько змеиный яд также являются протеазами клана PA, такими как гремучая змея гемотоксин и препятствовать каскаду свертывания крови жертвы. Кроме того, бактерии, такие как Золотистый стафилококк выделять эксфолиативный токсин которые переваривают и повреждают ткани хозяина. Многие вирусы выражают свои геном как единый массивный полипротеин и использовать протеазу клана PA, чтобы расщепить его на функциональные единицы (например, полиомиелит, норовирус, и Протеазы TEV ).[19][20]
Также есть несколько псевдоферменты в суперсемействе, где остатки каталитической триады были мутированы и, таким образом, функционируют как связывающие белки.[21] Например, гепарин -связывающий белок Азуроцидин имеет глицин вместо нуклеофила и серин вместо гистидина.[22]
Семьи
Внутри клана PA (P = протеазы смешанного нуклеофилы ) семейства обозначаются их каталитическим нуклеофилом (C =цистеиновые протеазы, S =сериновые протеазы ). Несмотря на отсутствие гомологии последовательностей для клана PA в целом, отдельные семьи внутри него могут быть идентифицированы по сходству последовательностей.
Смотрите также
Рекомендации
- ^ а б Ролингс Н.Д., Барретт А.Дж., Бейтман А. (январь 2012 г.). «МЕРОПС: база данных протеолитических ферментов, их субстратов и ингибиторов». Исследования нуклеиновых кислот. 40 (Проблема с базой данных): D343-50. Дои:10.1093 / nar / gkr987. ЧВК 3245014. PMID 22086950.
- ^ а б c d е Базан Дж. Ф., Флеттерик Р. Дж. (Ноябрь 1988 г.). «Вирусные цистеиновые протеазы гомологичны трипсиноподобному семейству сериновых протеаз: структурные и функциональные последствия». Труды Национальной академии наук Соединенных Штатов Америки. 85 (21): 7872–6. Bibcode:1988PNAS ... 85.7872B. Дои:10.1073 / pnas.85.21.7872. ЧВК 282299. PMID 3186696.
- ^ а б c d Ласкар А., Роджер Э. Дж., Чаттерджи А., Мандал С. (май 2012 г.). «Моделирование и структурный анализ сериновых протеаз клана PA». BMC Research Notes. 5: 256. Дои:10.1186/1756-0500-5-256. ЧВК 3434108. PMID 22624962.
- ^ Барбоса Дж. А., Салдана Дж. В., Гаррат Р. К. (июль 1996 г.). «Новые особенности активных сайтов сериновых протеаз и карманов специфичности: анализ последовательности и исследования моделирования глутамат-специфических эндопептидаз и эпидермолитических токсинов». Белковая инженерия. 9 (7): 591–601. Дои:10.1093 / белок / 9.7.591. PMID 8844831.
- ^ «МЕРОПС - протеазы архей S01».
- ^ Руис-Перес Ф, Натаро Дж. П. (март 2014 г.). «Бактериальные сериновые протеазы, секретируемые путем аутотранспортера: классификация, специфичность и роль в вирулентности». Клеточные и молекулярные науки о жизни. 71 (5): 745–70. Дои:10.1007 / s00018-013-1355-8. ЧВК 3871983. PMID 23689588.
- ^ а б c Buller AR, Townsend CA (февраль 2013 г.). «Внутренние эволюционные ограничения на структуру протеазы, ацилирование фермента и идентичность каталитической триады». Труды Национальной академии наук Соединенных Штатов Америки. 110 (8): E653-61. Bibcode:2013PNAS..110E.653B. Дои:10.1073 / pnas.1221050110. ЧВК 3581919. PMID 23382230.
- ^ де Хаен К., Нейрат Х, Теллер, округ Колумбия (февраль 1975 г.). «Филогения сериновых протеаз, связанных с трипсином, и их зимогенов. Новые методы исследования отдаленных эволюционных взаимоотношений». Журнал молекулярной биологии. 92 (2): 225–59. Дои:10.1016/0022-2836(75)90225-9. PMID 1142424.
- ^ Леск А.М., Фордхэм В.Д. (май 1996 г.). «Сохранение и изменчивость структур сериновых протеиназ семейства химотрипсинов». Журнал молекулярной биологии. 258 (3): 501–37. Дои:10.1006 / jmbi.1996.0264. PMID 8642605.
- ^ Горбаленя А.Е., Блинов В.М., Донченко А.П. (январь 1986 г.). «Кодируемая полиовирусом протеиназа 3C: возможная эволюционная связь между семействами клеточных сериновых и цистеиновых протеиназ». Письма FEBS. 194 (2): 253–7. Дои:10.1016/0014-5793(86)80095-3. PMID 3000829.
- ^ а б Phan J, Zdanov A, Evdokimov AG, Tropea JE, Peters HK, Kapust RB, Li M, Wlodawer A, Waugh DS (декабрь 2002 г.). «Структурные основы субстратной специфичности протеазы вируса травления табака». Журнал биологической химии. 277 (52): 50564–72. Дои:10.1074 / jbc.M207224200. PMID 12377789.
- ^ Аллер М., Черная М.М., Малькольм Б.А., Джеймс М.Н. (май 1994 г.). «Цистеиновые протеиназы пикорнавируса 3С имеют складку, аналогичную химотрипсиноподобным сериновым протеиназам». Природа. 369 (6475): 72–6. Bibcode:1994Натура 369 ... 72А. Дои:10.1038 / 369072a0. PMID 8164744.
- ^ Снайдер Э.Дж., Вассенаар А.Л., ван Динтен Л.С., Спаан В.Дж., Горбаленя А.Э. (март 1996 г.). «Протеаза артеривируса nsp4 является прототипом новой группы химотрипсиноподобных ферментов, сериновых протеаз 3С-подобных». Журнал биологической химии. 271 (9): 4864–71. Дои:10.1074 / jbc.271.9.4864. PMID 8617757.
- ^ Догерти В.Г., Паркс Т.Д., Кэри С.М., Базан Дж. Ф., Флеттерик Р. Дж. (Сентябрь 1989 г.). «Характеристика каталитических остатков протеиназы 49 кДа вируса травления табака». Вирусология. 172 (1): 302–10. Дои:10.1016/0042-6822(89)90132-3. PMID 2475971.
- ^ Ласкар А., Роджер Э. Дж., Чаттерджи А., Мандал С. (май 2012 г.). «Моделирование и структурный анализ сериновых протеаз клана PA». BMC Research Notes. 5 (1): 256. Дои:10.1186/1756-0500-5-256. ЧВК 3434108. PMID 22624962.
- ^ Шафи Т., Гатти-Лафранкони П., Минтер Р., Холлфельдер Ф. (сентябрь 2015 г.). «Эволюция по восстановлению инвалидности ведет к химически универсальной протеазе, допускающей нуклеофилы». ChemBioChem. 16 (13): 1866–1869. Дои:10.1002 / cbic.201500295. ЧВК 4576821. PMID 26097079.
- ^ Судзуки Ю., Сакамото Ю., Танака Н., Окада Н., Морикава Ю., Огасавара В. (март 2014 г.). «Идентификация каталитической триады экзопептидаз семейства S46, тесно связанных с эндопептидазами клана PA». Научные отчеты. 4: 4292. Дои:10.1038 / srep04292. ЧВК 3944710. PMID 24598890.
- ^ Sakamoto Y, Suzuki Y, Iizuka I, Tateoka C, Roppongi S, Fujimoto M, Inaka K, Tanaka H, Masaki M, Ohta K, Okada H, Nonaka T, Morikawa Y, Nakamura KT, Ogasawara W, Tanaka N (май 2014 г.) ). «Пептидазы S46 являются первыми экзопептидазами, входящими в клан PA». Научные отчеты. 4: 4977. Дои:10.1038 / srep04977. ЧВК 4021333. PMID 24827749.
- ^ Сальвесен Г (2013). Ролингс Н. (ред.). Справочник протеолитических ферментов. Бостон: Academic Press. ISBN 9780123822192.
- ^ Polgár L (октябрь 2005 г.). «Каталитическая триада сериновых пептидаз». Клеточные и молекулярные науки о жизни. 62 (19–20): 2161–72. Дои:10.1007 / s00018-005-5160-х. PMID 16003488.
- ^ Тодд А.Е., Оренго, Калифорния, Торнтон Дж. М. (октябрь 2002 г.). «Последовательность и структурные различия между ферментными и неферментными гомологами». Структура. 10 (10): 1435–51. Дои:10.1016 / s0969-2126 (02) 00861-4. PMID 12377129.
- ^ Иверсен Л.Ф., Каструп Ю.С., Бьёрн С.Е., Расмуссен ПБ, Виберг ФК, Флодгаард Х.Дж., Ларсен И.К. (апрель 1997 г.). «Структура HBP, многофункционального белка со складкой сериновой протеиназы». Структурная биология природы. 4 (4): 265–8. Дои:10.1038 / nsb0497-265. PMID 9095193.
внешняя ссылка
- МЕРОПЫ - Обширная база данных протеаз
- Надсемейство - База данных белковых складок


